MEJORA DE LAS PRUEBAS DE HER2 EN EL CÁNCER DE MAMA
Referencias bibliográficas:
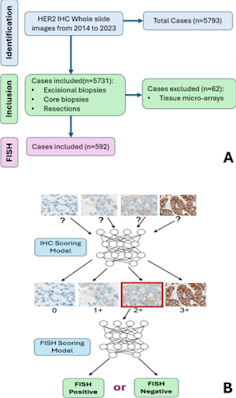
| 1. | Macaulay DO, Han W, Zarella MD, García CA, Tavolara TE. Mejora de las pruebas de HER2 en cáncer de mama: predicción de las evaluaciones de hibridación in situ con fluorescencia (FISH) a partir de imágenes de inmunohistoquímica mediante aprendizaje profundo. J Pathol Clin Res [Internet]. 2025;11(2):e70024. Disponible en: http://dx.doi.org/10.1002/2056-4538.70024 |
| 2. | Macaulay DO, Han W, Zarella MD, García CA, Tavolara TE. Mejora de las pruebas de HER2 en cáncer de mama: predicción de las evaluaciones de hibridación in situ con fluorescencia (FISH) a partir de imágenes de inmunohistoquímica mediante aprendizaje profundo. J Pathol Clin Res [Internet]. 2025;11(2):e70024. Disponible en: https://pubmed.ncbi.nlm.nih.gov/40050230/ Google.es. [citado el 15 de junio de 2025]. Disponible en: https://scholar.google.es/scholar?start=20&q=Enhancing+HER2+testing+in+breast+cancer&hl=es&as_sdt=0,5 |